Systembiotechnologie
Gruppe Systembiotechnologie
Die metabolischen Fähigkeiten von Mikroorganismen sind für den Übergang zu einer umweltfreundlichen und nachhaltigen chemischen Industrie essenziell. Bioprozesse mit Mikroben verlaufen bei niedrigen Temperaturen, geringem Energiebedarf und ohne chemische Abfälle zu erzeugen. Neben der technischen Nutzung ist der Stoffwechsel aber auch ein faszinierendes Netzwerk, das den Zellen Wachstum auch unter verändernden ökologischen Bedingungen ermöglicht. Der Stoffwechsel ist gleichzeitig robust und anpassungsfähig.
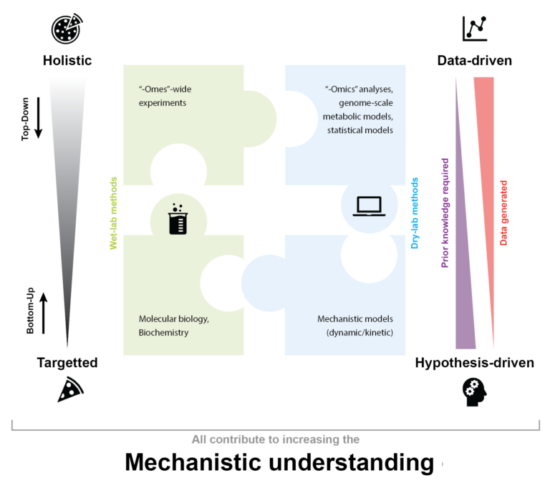
Wir erforschen und entwickeln den Stoffwechsel im Hinblick auf die Entwicklung industrieller biotechnologischer Lösungen, die Prozesse basierend auf fossilen Rohstoffen ersetzen. Wir nutzen dabei mathematische Modellierung, um die hochkomplexen Interaktionen in Stoffwechselnetzen zu verstehen, zu quantifizieren und zu modifizieren. Außerdem analysieren wir das Verhalten lebender Organismen auf verschiedenen Ebenen, um die Skalierung von Bioprozessen zu optimieren. Hier betrachten wir die Verwendung verschiedener Substrate und dynamische Kulturbedingungen.

Unsere Forschungsgruppe verfügt über ein breites Fachwissen im Bereich der Modellierung von Stoffwechselnetzwerken, einschließlich quantitativer Flussanalyse unter dynamischen Bedingungen unter Verwendung von 13C-Technologien [1, 2], der Integration zellulärer Ressourcen wie des Proteoms in dynamische Flussvorhersagen [3, 4] und der Integration thermodynamischer Randbedingungen [5]. Unsere experimentellen Möglichkeiten umfassen die Kultivierung in Bioreaktoren im Maßstab von 0,5 – 15 Litern unter dynamischen Bedingungen und definierten Fütterungsprofilen. Die Versuchsaufbauten ermöglichen schnelle Probenahmen in Verbindung mit hochauflösender Massenspektrometrie, um detaillierte metabolomische und proteomische Messungen durchzuführen.
Die nächste Generation von biotechnologischen Rohstoffen wird aus einer Kombination von Kohlenstoffquellen aus landwirtschaftlichen Abfällen (Rohstoffe der 2. Generation) und Stoffen, die aus der elektrochemischen Reduktion von CO2 stammen, wie Methan oder Methanol, bestehen. Augenblicklich betrachten wir die Dynamik und die Wechselwirkungen in Zellen unter Mehrsubstrat-Bedingungen. Die Verwendung von Stoffwechselmodellen zum Verständnis der Wechselwirkungen zwischen den verschiedenen Assimilationswegen sowie die Untersuchung des Stoffwechsel- und des Proteom-Verhaltens und der Reaktionen der Zelle unter verschiedenen statischen oder dynamischen Fütterungsprofilen wird es uns ermöglichen, Mikroorganismen zu entwickeln, die in der Lage sind, Mischungen von sich ergänzenden nachhaltigen Rohstoffen zu nutzen und so eine vollständig erneuerbare Produktion von Chemikalien zu ermöglichen.
Lehre:
Die Lehrtätigkeit der Gruppe ist in den Lehrplan des Studiengangs Life Science Engineering (LSE) an der FAU Erlangen-Nürnberg eingebettet. Die Studierenden tauchen ein in die spannende Welt der metabolischen Netzwerke. Studenten erlernen die Prozesse in Zellen zu abstrahieren, Modelle zu erstellen und zu untersuchen und lernen die neuesten Ansätze der Systembiologie kennen. Alle Vorlesungen werden durch computergestützte Übungen und Praktika ergänzt, in denen die Studierenden die erlernten Methoden direkt anwenden. Die Kurse umfassen:
– Metabolic Engineering, 5 ECTS im LSE-Bachelorprogramm.
– Systembiotechnologie VT, 7.5 ECTS im LSE-Masterstudiengang
– Teile der Bioanalytik, 5 ECTS im LSE-Bachelorstudiengang
Darüber hinaus sind wir aktiv in der Gestaltung und Lehre von Post-Master-Kursen:
– Integrated Multi-Omics approaches for Improvement of Industrial Microbes (BioTech Delft, see Biotechnology Courses.nl)
Referenzen:
- Suarez-Mendez CA, Hanemaaijer M, ten Pierick A, Wolters JC, Heijnen JJ, Wahl SA: Interaction of storage carbohydrates and other cyclic fluxes with central metabolism: A quantitative approach by non-stationary <sup>13</sup>C metabolic flux analysis. Metabolic Engineering Communications 2016, 3.
- Verhagen KJ, van Gulik WM, Wahl SA: Dynamics in redox metabolism, from stoichiometry towards kinetics. Current Opinion in Biotechnology 2020, 64.
- Verhagen KJA, Eerden SA, Sikkema BJ, Wahl SA: Predicting Metabolic Adaptation Under Dynamic Substrate Conditions Using a Resource-Dependent Kinetic Model: A Case Study Using Saccharomyces cerevisiae. Frontiers in Molecular Biosciences 2022, 9.
- Paez-Watson T, van Loosdrecht MCM, Wahl SA: Predicting the impact of temperature on metabolic fluxes using resource allocation modelling: Application to polyphosphate accumulating organisms. Water Res 2023, 228:119365.
- Cueto-Rojas HF, van Maris AJA, Wahl SA, Heijnen JJ: Thermodynamics-based design of microbial cell factories for anaerobic product formation. Trends in Biotechnology 2015, 33.